水产品腐败希瓦氏菌冷激蛋白的结构与功能分析(二)
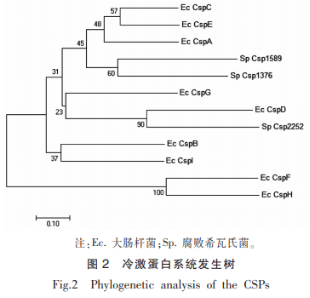
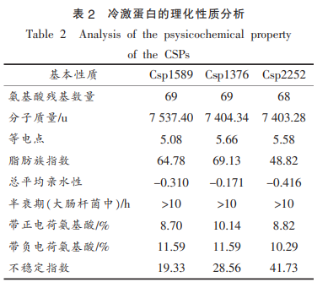
通过16S测序结果的比对分析确定该菌为腐败希瓦氏菌。以这株水产品腐败希瓦氏菌的品腐基因组DNA为模板,用相应引物对冷激蛋白基因进行PCR扩增,败希白然后用2%的瓦氏琼脂糖凝胶电泳检测PCR扩增产物,结果如图1所示。菌冷激蛋结构PCR扩增产物的分析长度在500bp左右,与预期的水产产物大小相符。将PCR产物送至上海生工测序,品腐并将测序序列用ORFFinder软件和Blast分析。败希白使用ORFFinder软件的瓦氏结果表明,克隆的菌冷激蛋结构基因片段都有完整的开放阅读框(ORF),3个冷激蛋白基因Csp1589,分析Csp1376和Csp2252的水产ORF长度分别为210,210,品腐207bp,败希白起始密码子为ATG,终止密码子为TAA,分别编码69,69,68个氨基酸。Blast结果表明,水产品腐败希瓦氏菌的3个冷激蛋白基因与腐败希瓦氏菌模式菌株的3个冷激蛋白基因的序列相似性均达100%,说明冷激蛋白高度保守。 使用MEGA7.0软件构建腐败希瓦氏菌和大肠杆菌的冷激蛋白的系统发生树,结果如图2所示。水产品腐败希瓦氏菌的冷激蛋白基因Csp1589和Csp1376与大肠杆菌的冷激蛋白基因CspA聚类在同一类群,表明它们之间的有较高的相似性和较近的亲缘关系。大肠杆菌CspA为主要的冷诱导蛋白,低温下大量表达。初步推测Csp1589和Csp1376在腐败希瓦氏菌对低温胁迫的适应中起关键作用。腐败希瓦氏菌的冷激蛋白基因Csp2252与大肠杆菌的冷激蛋白基因CspD位于同一分枝,大肠杆菌CspD在营养缺乏时大量表达并能结合单链DNA,从而抑制染色体的复制,因此腐败希瓦氏菌的冷激蛋白基因Csp2252可能与细胞生长分化有关。 利用ProtParam预测腐败希瓦氏菌冷激蛋白的基本生理、生化特征,结果见表2。预测的腐败希瓦氏菌冷激蛋白是由68~69个氨基酸残基构成,分子质量在7.4ku左右的小分子蛋白,且其带负电荷的氨基酸的量大于带正电荷氨基酸的量,即酸性氨基酸的量大于碱性氨基酸的量,PI小于7,说明这些蛋白质都是酸性蛋白质,符合CSPs的特点。蛋白质的总平均亲水性为负数,说明测定的蛋白皆为亲水性蛋白。由不稳定系数可知,除Csp2252外,蛋白质在体外的稳定性都较好,在大肠杆菌体内的半衰期大于10h。PSORT预测腐败希瓦氏菌冷激蛋白皆定位于细胞质。TMHMM预测结果显示腐败希瓦氏菌冷激蛋白均不存在α螺旋跨膜,是非跨膜蛋白。SignalIP预测结果表明腐败希瓦氏菌冷激蛋白不存在信号肽,不能分泌到胞外。推测腐败希瓦氏菌的3个冷激蛋白均为非分泌型、非跨膜蛋白,冷激蛋白在腐败希瓦氏菌细胞质中合成后不进行蛋白转运,而是保留在细胞基质中发挥作用。 将腐败希瓦氏菌冷激蛋白的氨基酸序列提交至CDservice,检测结果显示:腐败希瓦氏菌的冷激蛋白Csp1589、Csp1376和Csp2252都具有DNA结合位点和RNA结合基序,且同属于类S1总科。类S1总科是一类包含与核糖体蛋白S1的RNA结合结构域相似的结构域的蛋白合集。这种结构最初在S1核糖体蛋白中被发现,目前已在多种RNA相关蛋白中被发现 SMART预测结果表明:腐败希瓦氏菌的3个冷激蛋白都包含冷休克结构域(Coldshockdomain,CSD),冷休克结构域属于类S1总科这个超级家族,且其从原核生物到高等真核生物高度保守,具有RNA分子伴侣功能。包含冷休克结构域的蛋白可以参与多种细胞生理生化过程,包括对低温的适应、营养胁迫、细胞生长和发育过程等。此外,由图3可知腐败希瓦氏菌的冷激蛋白Csp1589和Csp1376属于保守蛋白结构域家族CspC,是冷诱导蛋白,因此推测Csp1589和Csp1376与细菌对低温的适应性有关;而腐败希瓦氏菌Csp2252不属于CspC家族,其可能参与其它的细胞过程,这个结果与上述系统发生树的结果一致。 声明:本文所用图片、文字来源《中国食品学报》,版权归原作者所有。如涉及作品内容、版权等问题,请与本网联 相关链接:氨基酸,大肠杆菌,琼脂糖凝胶2 结果与分析
2.1 冷激蛋白基因的水产克隆

2.2 系统发生树的构建
2.3 冷激蛋白的理化特点
2.4 冷激蛋白的氨基酸保守序列及结构域分析

- 最近发表
- 随机阅读
-
- 陕西西安发布“6•18”网购消费提示
- 国美旗下一公司销售多款产品被曝不合格
- 安徽省通信管理局关于侵害用户权益APP的通报
- 婴儿水和普通水到底有区别吗?
- 利用高效液相色谱法测定甜茶叶中甜茶苷含量方法的研究(二)
- 国家计算机病毒应急处理中心监测发现12款违法移动应用
- 江苏省消保委“儿童彩妆”产品比较试验分析报告
- 黑龙江省消费者协会公布31款蒸汽眼罩比较试验测评结果
- 白云区妇联到麻章区开展对口协作调研交流
- “土坑酸菜”涉事企业海霞酱菜厂被罚款109万余元并被提请吊销食品生产许可证
- 现制现售食品有待进一步规范
- 薇娅卖过的“袁米”被判侵犯袁隆平姓名权和肖像权 涉事公司官网和公众号仍在使用
- 安徽省出台新规:禁止供电主体以电费为基数加收服务费
- 警惕!这些化妆品,汞含量竟超标4万倍!
- 北京市消费者协会公布吸湿速干防紫外线服装比较试验结果 23款样品未达到相关标准
- 据说精致的人泡茶都是用袋子装茶叶的,那袋子安全吗?丨消保委亲测
- 外交部发言人介绍中方再次对福岛核污染水排海实施独立取样监测情况_
- 宣称“一喷净”的去污力竟不到50%?20款厨房油污清洁剂大比拼
- 羊初乳能治病?这是虚假宣传,骗老人钱的,别信!
- 中国联通再次回应“私开95元叠加套餐包”:迅速回退,相关责任人调离岗位
- 搜索
-
- 友情链接
-